1. PCR (Polymerase chain reaction)
2. Clonación
3. Las enzimas de restricción
4. DNA fingerprinting (Huella de adn)
5. ADN Recombinante
1.PCR (Polymerase chain reaction)
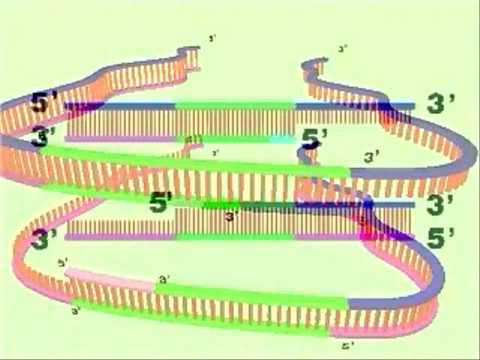
La Reacción en Cadena de la Polimerasa (PCR) es una técnica "in vitro" que imita la habilidad natural de la célula de duplicar el ADN.
Se trata de una técnica usada para crear un gran número de copias de un segmento de ADN, que utiliza ciclos de desnaturalización, apareamiento con cebadores y extensión por una ADN polimerasa termoresistente.
Hasta la década de 1980, el único método para obtener grandes cantidades de un fragmento de ADN era clonándolo en vectores adecuados e introduciéndolo y multiplicándolo en bacterias. En el año 1985, un investigador norteamericano, Kary Mullis (acreedor del Premio Nobel en Química 1993 por este aporte), desarrolló un método que permite, a partir de una muestra muy pequeña de ADN, obtener millones de copias de ADN in vitro, en unas pocas horas y sin necesidad de usar células vivas. Esta técnica, llamada reacción en cadena de la polimerasa (PCR), requiere conocer la secuencia de nucleótidos de los extremos del fragmento que se quiere amplificar. Estas secuencias se usan para diseñar dos oligonucleótidos sintéticos de ADN complementarios a una porción de cada una de las dos cadena de la doble hélice.
1. La mezcla de reacción contiene la secuencia de DNA que se quiere amplificar, dos oligonucleótidos sintéticos (P1 y P2) que servirán como cebadores, una DNA polimerasa termoestable (Taq) y los cuatro desoxirribonucleótidos trifosfato –dATP, dGTP, dCTP y dTTP–.
2. Proceso:
La mezcla de reacción se somete a ciclos sucesivos, cada uno correspondiente a una fase de desnaturalización, una de hibridación o alineación y una de elongación.
a) Durante la desnaturalización, que se realiza por calentamiento de la mezcla a 95ºC, se separan las dos cadenas del ADN molde.
b) Durante la hibridación, la temperatura de incubación se reduce para permitir el apareamiento de las bases de ambos cebadores en el sitio donde encuentran una secuencia complementaria.
c) Durante la fase de elongación, la mezcla se calienta a 72ºC y la enzima Taq ADN polimerasa se usa para replicar las hebras de DNA. La Taq polimerasa comienza el proceso de extensión de la cadena complementaria a partir del extremo 3’ de los cebadores. Al finalizar cada ciclo, la cantidad de ADN molde disponible para el ciclo siguiente aumenta al doble.
Entre muchas de las aplicaciones que la PCR pone a disposición se encuentran la detección precoz o prenatal de enfermedades genéticas, la detección de infecciones virales latentes o la producción de grandes cantidades de fragmentos de ADN humano a una velocidad muy superior a la posible mediante otros métodos. Esta técnica también se aplica para estudios de identidad y filiación.
PCR a partir de ARN.
El genoma de muchos virus de importancia clínica está compuesto de ARN en lugar de ADN, los más sobresalientes son el Virus de la Inmunodeficiencia Humana (VIH), el Virus de la Hepatitis C (HCV) y la familia de Enterovirus (EV).
Transcripción Reversa- PCR (RT-PCR).
Dado que el ARN usualmente es de una sola hebra y es sensible al calor, es necesario hacer una transcripción reversa (RT) antes de iniciar la amplificación por PCR. La transcripción reversa genera una copia de la hebra de RNA, pero esta copia es ADN complementario (cADN) el cual es estable al calor y puede resistir la metodología PCR.
Los pasos de la RT-PCR son:
Transcripción reversa: Unión del partidor a la secuencia de ARN objetivo.
Transcripción reversa: La polimerasa rTth cataliza la extensión del partidor mediante la incorporación de nucleótidos complementarios.
Fin de transcripción reversa, se obtiene la hebra del ADN complementario al ARN.
2.Clonación

Recordemos que los plásmidos son moléculas de ADN circulares, pequeñas, que se encuentran
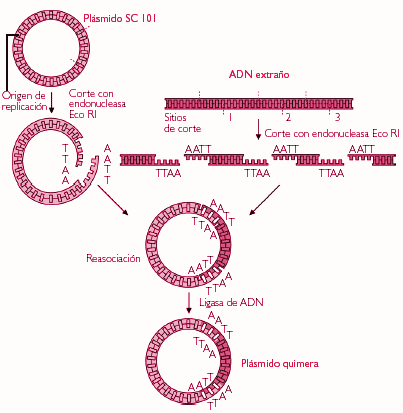
en las bacterias por fuera del ADN cromosómico.Dado que se adaptan a albergar otro pedazo de ADN, se abre la posibilidad de poner un gen determinado (o un tramo de ADN determinado) en un plásmido circular, generando un ADN recombinante. En estas circunstancias el plásmido está funcionando como un “vector”: es capaz de llevar el trozo de ADN (que denominamos inserto) mantenerlo, tenerlo aislado, lo cual era uno de los propósitos de la manipulación genética.
El plásmido, que naturalmente está en las bacterias, puede volver a las bacterias y crecer con ellas. Se pueden transformar bacterias con plásmidos que llevan inserto. Una vez dentro de la bacteria, el plásmido se replica con ella. Así se consigue que el trozo de ADN además de estar
aislado, sea amplificado y se obtienen rápidamente muchas copias idénticas. Decimos que el trozo de ADN fue clonado. Teniendo una cantidad mayor de ADN del tramo de interés éste puede ser secuenciado, cortado con enzimas de restricción u otras manipulaciones.
3.Las enzimas de restricción

Uno de los avances más importante en los inicios de la biología molecular, fue el descubrimiento de las endonucleasas de restricción, es decir de enzimas que pueden cortar el ADN y que tienen la ventaja de que lo cortan en sitios concretos. Fueron descubiertas en bacterias, y son enzimas que estas bacterias usan para destruir ADN que ingresa a ellas, por ejemplo ADN de virus bacteriófagos. Con las enzimas, la bacteria puede degradar este ADN foráneo sin degradar su propio ADN. Varias de ellas fueron identificadas en la década del 70 y siguieron descubriéndose otras posteriormente, aisladas de diferentes cepas bacterianas. La ventaja de estas enzimas es que reconocen un sitio para cortar, pueden ser un diseño de 4, 6, 8 bases, pero tienen que estar
organizadas con una secuencia exacta y no otra. Por ejemplo, la enzima que se llama EcoR1 (porque se aisló de la bacteria Escherichia coli, que reside normalmente en el intestino), solo corta si encuentra la secuencia GAATTC. Si hay una base que está cambiada ya no corta. Estas enzimas de restricción, entonces permiten cortar el ADN en sitios especìficos. Algunas de ellas cortan dejando extremos cohesivos que se pueden pegar nada más que por complementariedad de bases. Esto quiere decir que un extremo que generó una enzima y otro que generó la misma enzima aunque provengan de moléculas de ADN diferentes, se pueden unir y generar lo que se llamó
inicialmente un ADN recombinante. Este hito de los años 70 de poder recombinar, es decir hacer construcciones genéticas, hacer ingeniería en genética fue la base de una enormidad de otros avances. En 1972 justamente se genera el primer ADN recombinante combinando el ADN de un plásmido, con un tramo de ADN de anfibio.
4.Dna fingerprinting (Huella de adn)

La tecnología denominada huella de ADN (DNA fingerprinting) permite comparar muestras de ADN de diversos orígenes, de manera análoga a la comparación de huellas dactilares. En esta técnica los investigadores utilizan también las enzimas de restricción para romper una molécula de ADN en pequeños fragmentos que separan en un gel al que someten a una corriente eléctrica (electroforesis); de esta manera, los fragmentos se ordenan en función de su tamaño, ya que los más pequeños migran más rápidamente que los de mayor tamaño. Se puede obtener así un patrón de bandas o huella característica de cada organismo. Se utiliza una sonda (fragmento de ADN marcado) que hibride (se una específicamente) con algunos de los fragmentos obtenidos y, tras una exposición a una película de rayos X, se obtiene una huella de ADN, es decir, un patrón de bandas negras característico para cada tipo de ADN.
El DNA Fingerprint es una técnica utilizada para saber si una muestra de DNA pertenece a cierta persona. Al igual que una huella digital el DNA de cada persona es único, o mejor dicho, la secuencia de los pares de bases del DNA es única para cada persona. Científicos son capaces de identificar a una persona solamente por el ordenamiento de los pares de bases de su DNA. El DNA Fingerprint es usado en casos de paternidad y maternidad para identificar a los padres de un niño. Otra aplicación es en la medicina forense en casos de identificación de víctimas y establecer la inocencia o culpabilidad de los sospechosos. Y una tercera aplicación sería como identificación personal, pero por el momento el Fingerprint es muy complejo y costoso para usarse como identificación personal.
El DNA Fingerprint requiere la utilización de otras técnicas. El Southern blot es utilizado para el Fingerprint ya que con el uso de una sonda radioactiva es posible identificar fragmentos de una secuencia de DNA. Otra técnica usada es el PCR. Con esta técnica es posible amplificar el DNA de una pequeña muestra. El PCR es una técnica necesaria para casos criminales ya que el DNA encontrado en la escena del crimen generalmente no es suficiente para poder trabajar con él.
5.ADN Recombinante
A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería
genética o lo que se ha llamado técnicas del ADN recombinante. Y esto ocurrió, en comparación con lo que fue el resto de la historia de la ciencia, de forma muy rápida entre los años 70 y 80.
En estas primeras etapas se estaba trabajando sobre la posibilidad de manipular los genes, es decir:
A) tenerlos aislados,
B) amplificarlos, en el sentido de tener muchas copias de la misma secuencia,
C) conocer la secuencia exacta, es decir el orden de las bases de esos genes
D) una vez aislado poderlo expresar fuera de su localización natural, lo cual tendrá una enormidad de otras aplicaciones.
Toda esta manipulación genética está simplemente basada en unas pocas propiedades del ADN que han permitido avanzar muchísimo en las técnicas. Recordemos: el hecho de que el ADN sea una doble cadena, y las cadenas sean complementarias y que la complementariedad de bases sea un requisito suficiente para que dos cadenas que estaban en simple hebra se encuentren y se vuelvan a reconstituir es la base de la mayor parte de la manipulación. Dos simples cadenas de ADN reconstituyen una doble cadena unida por puentes de hidrógeno basado simplemente en la complementariedad de bases, en el hecho de que si en una de las hebras hay una serie de nucleótidos con las bases GCAT cualquier otra hebra que tenga CGTA, es decir complementaria, va
a poder unirse y reconstituir una doble cadena en determinadas condiciones de temperatura y de pH
dadas. Esta es una de las características básicas. A esto se agrega el hecho de que el ADN tiene la
información en el orden de las bases y que el código por el cual esa información es trascripta y
traducida a una proteína es prácticamente universal: ese tramo de ADN si es que codifica para algo
puede producir esa proteína en diversas condiciones.
graciasss me sirvió bastante la información
ResponderBorrarGracias por el post, es de mucha ayuda.. Saludos..
ResponderBorrar